2022年2月2日 14:00
生物は環境変化に応答して遺伝子発現を変化させます。バクテリアではシグマ因子と呼ばれるタンパク質を使い分けることで発現する遺伝子を決定します。光合成を行うバクテリアであるラン藻は糖の分解経路の遺伝子発現を行うシグマ因子としてSigEが存在します。
明治大学農学部農芸化学科環境バイオテクノロジー研究室の仮屋園 遼 研究推進員と小山内 崇 准教授との研究グループは、ラン藻のモデル生物であるシネコシスティス注1(学名Synechocystis sp.)のSigEについて、ChIP-seqという手法を用いてゲノム上の結合箇所を特定しました。
■ 研究成果のポイント
●ラン藻の糖分解やバイオプラスチック生産に関与する遺伝子群を制御するシグマ因子SigEの結合領域をゲノムワイドに特定した。
●得られたSigEの結合領域の情報からSigE特異的に転写される遺伝子のプロモータに必要な要素を推定し、実験的に証明した。
●今回得られた情報を利用することで、将来的にラン藻の遺伝子の発現を大規模に改変・制御できる可能性がある。
■ 要旨
生物は環境が変化すると転写する遺伝子を変えて対応します。原核生物のバクテリアではシグマ因子と呼ばれるタンパク質が転写する遺伝子を選択しており、複数種類のシグマ因子を使い分けることで環境変化に対応した遺伝子を働かせます。
シグマ因子は遺伝子のプロモータ領域のDNA配列に結合することで転写する遺伝子を選択しています。生存に必須な遺伝子群(ハウスキーピング遺伝子)の転写は主要シグマ因子と呼ばれるシグマ因子が担い、環境変化に応答する遺伝子群は代替シグマ因子と呼ばれるシグマ因子が転写します。
ラン藻は光合成を行うバクテリアで、光合成により二酸化炭素を同化して糖に変換しグリコーゲンとして貯蔵しますが、光合成のできない状況ではグリコーゲンおよび糖を分解して生命活動に必要なエネルギーを獲得します。研究チームの所属する研究室では淡水性単細胞モデルラン藻であるシネコシスティス(注1)(学名Synechocystis sp.)の糖分解経路を利用して、バイオプラスチック原料となる有機酸を生産させる研究を行なっています。
シネコシスティスではグリコーゲンと糖の分解を行う酵素を発現させるために、SigEという代替シグマ因子が働き、ハウスキーピング遺伝子の転写を担う主要シグマ因子としてSigAが働きます。しかしSigEがどのように糖の分解に関連する遺伝子群を選択して転写しているのかが不明でした。そこで今回研究チームはSigEとSigAがシネコシスティスのゲノムのどこに結合しているかを調べました、するとSigEの結合領域の8割はSigAと同じでハウスキーピング遺伝子に結合していましたが、SigEが転写する糖の分解に関連する遺伝子群ではSigEのみが結合し、SigAが結合していないことを明らかにしました。また、SigEの結合領域からSigEが転写する遺伝子を識別するためのDNA配列の要素を決定しました。
この研究は、明治大学農学部農芸化学科環境バイオテクノロジー研究室の小山内崇 准教授と仮屋園遼 研究推進員によって行われ、JST戦略的創造研究推進事業先端的低炭素化技術開発ALCA,科研費基盤Bの援助により行われました。本研究成果は、2022年1月29日発行の英国科学誌「The Plant Journal」のオンライン版に掲載されました。
【研究グループ】
明治大学 農学部農芸化学科
環境バイオテクノロジー研究室
准教授 小山内 崇(おさない たかし)
研究推進員 仮屋園 遼(かりやぞの りょう)
■ 1.背景
ラン藻は光合成を行う真正細菌で、海洋、淡水、陸上など幅広い環境に分布しています。ラン藻を含め、光合成を行う生物は光合成のできる日中と、できない夜間という、1日の中で非常に大きな環境変化に遭遇します。
生命活動は環境変化に応じて生存に最適な調節を受けます。生物は様々な調節方法を持っており、環境の変化に対応して酵素または酵素のネットワーク(代謝)が活性を調節したり、遺伝子の転写と翻訳を調節して酵素の量を変動させたりします。これらの調節方法の中で、転写の調節は他よりも長期の環境変化への対応を担います。
転写はRNAポリメラーゼと呼ばれるタンパク質の複合体が行います。ラン藻を含む真正細菌では、複合体のサブユニットの一つであるシグマ因子が遺伝子上流のプロモータ領域を認識して結合することで目的の遺伝子が転写されます。また、プロモータ領域には転写因子と呼ばれるタンパク質も結合し、RNAポリメラーゼと協調して転写を促進、あるいは抑制します。
環境変化に対応するための主要な手段として、真正細菌は複数のシグマ因子を使い分けています。生育に最適な環境下では主要シグマと呼ばれるシグマ因子が働き、ハウスキーピング遺伝子のプロモータを認識して転写します。一方で、最適ではない環境、例えば高温、栄養飢餓、そしてラン藻の場合光条件の変化などの状況下では、代替シグマと呼ばれるシグマ因子が働き、環境に適応するための遺伝子のプロモータを認識して転写します。
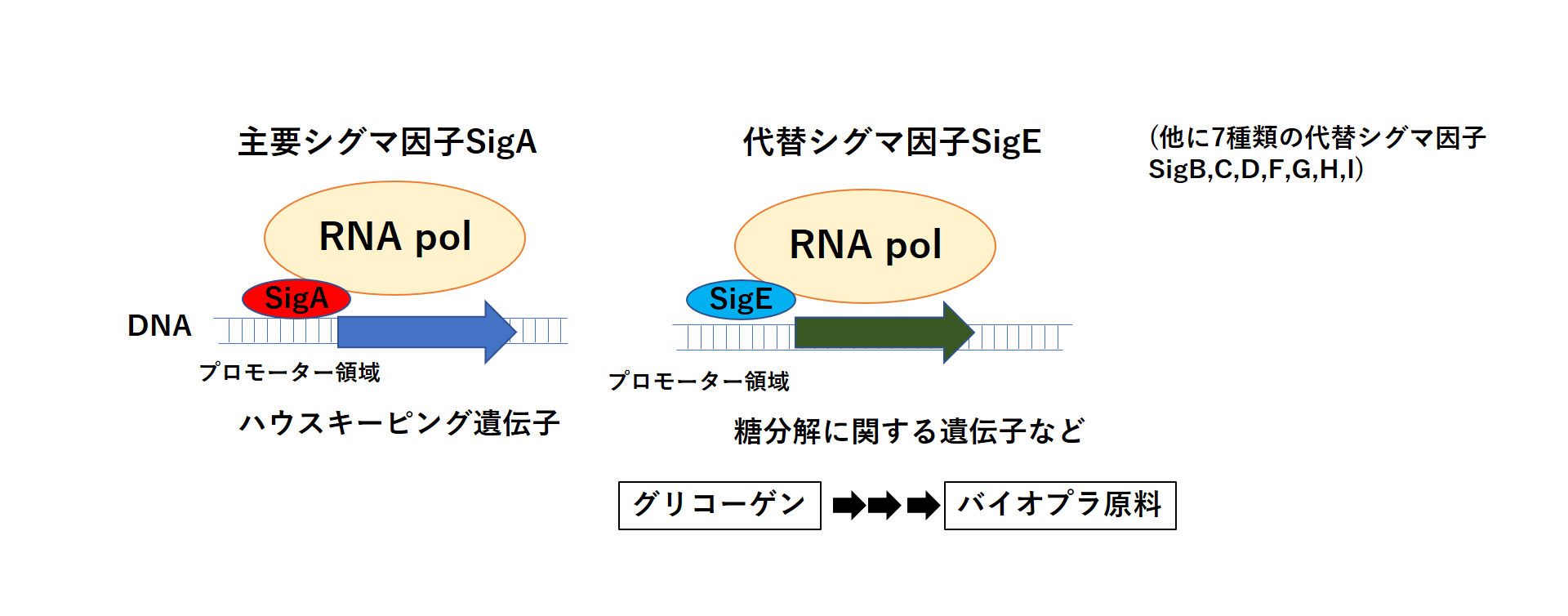
単細胞性ラン藻のモデル生物シネコシスティスは主要シグマ因子(SigA)のほか、8種類の代替シグマ因子を持っています。私たちの研究グループは代替シグマ因子の一つ、SigEに着目して研究を行ってきました。以前の研究では遺伝子操作によりSigE遺伝子を破壊したシネコシスティス細胞の遺伝子発現を調べ、SigEによって転写される遺伝子群(SigEのレギュロン)の中に糖の分解やバイオプラスチック生産に関連する遺伝子群を多く含むことを発見しています。また、その後の研究でSigEを過剰に働かせると、糖の分解やバイオプラスチック原料生産が促進されることもわかっていました。(図1)
図1. ハウスキーピング遺伝子の転写を担う主要シグマ因子SigAと、グリコーゲンおよび糖分解の遺伝子の転写を担う代替シグマ因子SigE
しかしこれまでの研究からは、SigEがどのような配列のプロモータを認識して転写を開始させるのかを決定することができませんでした。そこでSigEとSigAがゲノム上のどこに結合するかをクロマチン免疫沈降(ChIP)と呼ばれる手法により同定しました。これによりSigEが特異的に認識するプロモータ配列を明らかになり、SigEが固有の遺伝子群の転写を促進できる分子基盤を解明しました。
■ 2.研究手法と成果
シグマ因子が遺伝子のプロモータに結合することでRNAポリメラーゼによる転写が開始されます。そこで研究グループはシネコシスティスのゲノム上で、SigEがどこに結合しているかを調べる為のクロマチン免疫沈降シーケンス(ChIP-seq)実験を行いました。具体的には以下の操作を行います。(図2a)
図2. a ChIP-seq法の概略図 / b ChIP-seq法でわかったSigEおよびSigAの結合場所。横軸はシネコシスティスゲノム上の位置。縦軸はSigEまたはSigAのゲノム領域に対する結合強度
1. 遺伝子操作によりSigEにタグ配列注2を付加したシネコシスティスを作製する
2. 作製したシネコシスティスのDNA と、DNAに結合するタンパク質を薬剤で架橋する
3. 細胞を破壊し、DNAを200bp程度の長さにせん断する
4. タグ配列に特異的に結合する抗体ビーズを使ってSigEと架橋されたDNAを集める
5. 集めたDNAを精製して次世代シーケンサーで配列を読む
6. 読まれた配列からSigEが結合する領域を特定する
また、主要シグマ因子SigAについても同様にChIP-seqを行い、SigEの結合箇所と比較を行いました。SigEとSigAのChIP-seq実験により以下のことがわかりました。(図2b)
1. SigEもSigAもほとんどが遺伝子のプロモータ(転写開始点の近く)に分布する
2. SigEの結合場所478箇所のうち、394箇所はSigAの結合場所とオーバーラップしており、その場所は主にハウスキーピング遺伝子のプロモータである
3. SigEのレギュロンのプロモータはSigEのみが結合し、SigAが結合していないものが多い
これらの結果から“SigEとSigAはどちらもハウスキーピング遺伝子のプロモータを認識できるが、SigEのみが認識できるプロモータが存在し、SigEレギュロンのプロモータとして働く”という結論が導かれました。この結論は“SigEのみに転写される遺伝子群がある”という先行研究と“精製したSigAとSigEはどちらもハウスキーピング遺伝子を転写できる”という先行研究の両方を矛盾なく説明できます。
さらに研究チームはSigEレギュロンのSigE結合部分の配列から、SigEによる転写に必要なDNA配列の要素(コンセンサス配列)を決定しました。(図3a)コンセンサス配列が実際にSigE特異性に重要かを調べるため、シネコシスティスの細胞内でSigE存在下と非存在下でプロモータの強さを測る実験を行いました。(図3b)
図3. a ChIP-seqアッセイからわかったプロモータのコンセンサス配列。上段:SigA、SigEの結合するハウスキーピング遺伝子プロモータのコンセンサス配列。下段:SigEに転写される遺伝子プロモータのコンセンサス配列。 bプロモータアッセイの概略図 c 配列を改変したプロモータによるプロモータ活性の変化。上段:SigEによって転写されるプロモータのコンセンサス配列をハウスキーピング遺伝子のものに改変し、SigEの有無にかかわらず転写されるようになった。中段:ハウスキーピング遺伝子のコンセンサス配列をSigE依存のプロモータのものに改変し、SigEの存在に依存して活性が上がるプロモータになった。
下段:別のハウスキーピング遺伝子のコンセンサス配列をSigE依存のプロモータのものに改変すると、SigEの有無にかかわらずほとんど活性が見られなくなった。
SigEによって転写される遺伝子のプロモータのコンセンサス配列をハウスキーピング遺伝子の物に改変したところ、SigEの有無に関係なく発現するようになりました。また逆に、ハウスキーピング遺伝子のプロモータについてコンセンサス配列をSigEによって転写される遺伝子のものに改変を試みました。すると、あるハウスキーピング遺伝子プロモータではSigEに依存した転写が起きるようになり、別のハウスキーピング遺伝子プロモータではSigEの有無にかかわらずほとんど転写が起きなくなりました。(図3c)二者の違いは上流に結合する転写因子の有無ではないかと推測されます。
これらの結果からSigEとSigAは基本的に同じプロモータを認識するが、微妙に異なるプロモータの”好み”の違いがSigEレギュロンのプロモータを生み出すことがわかりました。さらに、SigEが認識するプロモータ領域の上流に結合する転写因子がSigEを補助している可能性が示唆されました。(図4)
図4. SigAとSigEおよびハウスキーピング遺伝子とSigEに転写される遺伝子のプロモータの概念図
■ 3.今後の期待
研究チームは、シグマ因子SigEとSigAの結合箇所をChIP-seq法を用いてゲノムワイドに同定しました。シネコシスティスには他に7種類のシグマ因子があり、それぞれ異なる環境変化に対応した遺伝子群を発現させます。本研究の手法により他のシグマ因子についても結合箇所を同定し、シネコシスティスのシグマ因子による転写制御の網羅的な理解につながると期待されます。
転写制御はシグマ因子による制御だけでなく、様々な転写因子が遺伝子の転写を促進または抑制します。今回の研究で転写因子がSigEの転写を補助している可能性が示唆されたので、“どのような転写因子がどのシグマ因子を補助してどの遺伝子を制御するか”を調べることでよりシネコシスティスの転写制御の理解を深められると期待されます。
研究チームの所属する研究室ではシネコシスティスの代謝を改変し、光合成で作られた糖から有用物質を生産させる研究も行っています。SigEのような糖の分解酵素遺伝子の転写をグローバルに制御する因子の研究は、シネコシスティスによる物質生産の基礎となる知見が得られます。また、ラン藻にはシネコシスティス以外にも様々な物質生産上の利点を持った生物種がおり、それらの転写制御改変による物質生産においても役立つ知見となるでしょう。
■ 4.論文情報
<タイトル>
Identification of genome-wide distribution of cyanobacterial group 2 sigma factor SigE accountable for its regulon
(ラン藻のグループ2シグマ因子SigEにおけるゲノムワイドな分布の特定とSigEレギュロンの対応)
<著者名> Ryo Kariyazono, Takashi Osanai
<雑 誌> The Plant Journal
< DOI > 10.1111/tpj.15687
■ 5.補足説明
注1)シネコシスティス
淡水性、単細胞性のラン藻。全生物の中で3番目、ラン藻の中では最初に全ゲノムが決定されたモデル生物であり、シグマ因子の研究も進んでいる。
注2)タグ配列
短いポリペプチド鎖からなる配列で、特定の抗体と非常に高い特異性で結合する。タグ配列を目的のタンパク質に遺伝子操作により繋げることで、目的のタンパク質を抗体によって効率よく回収することができるようになる。










![[Amazon限定ブランド]CCL い・ろ・は・すラベルレス 2LPET ×8本 ミネラルウォーター 無味 製品画像:2位](https://m.media-amazon.com/images/I/41h0MHfvhkL._SL160_.jpg)






























![フィリップス(ディスプレイ) 221S9A/11 [21.5型液晶ディスプレイ/1920×1080/HDMI、D-Sub/スピーカー:あり/5年間フル保証] 製品画像:23位](https://thumbnail.image.rakuten.co.jp/@0_mall/etre/cabinet/itemimage28/1267245.jpg?_ex=128x128)








![週刊少年マガジン 2026年13号[2026年2月25日発売]【電子書籍】[ 久世蘭 ] 製品画像:29位](https://thumbnail.image.rakuten.co.jp/@0_mall/rakutenkobo-ebooks/cabinet/6646/2000019776646.jpg?_ex=128x128)
![異世界クラフトぐらし〜自由気ままな生産職のほのぼのスローライフ〜(コミック) : 9【電子書籍】[ 立山鈴 ] 製品画像:28位](https://thumbnail.image.rakuten.co.jp/@0_mall/rakutenkobo-ebooks/cabinet/5710/2000019775710.jpg?_ex=128x128)
![大河の一滴 最終章 [ 五木 寛之 ] 製品画像:27位](https://thumbnail.image.rakuten.co.jp/@0_mall/book/cabinet/5293/9784344045293_1_3.jpg?_ex=128x128)
![GQ JAPAN(ジーキュー ジャパン)特別表紙版 2026年 4月号 [雑誌] 製品画像:26位](https://thumbnail.image.rakuten.co.jp/@0_mall/book/cabinet/0464/4912045920464.gif?_ex=128x128)
![こうやって、僕は戦い続けてきた。 「理想の自分」に近づくための77の習慣 [ 菊池 雄星 ] 製品画像:25位](https://thumbnail.image.rakuten.co.jp/@0_mall/book/cabinet/0602/9784569860602_1_6.jpg?_ex=128x128)
![夏目友人帳 33 (花とゆめコミックス) [ 緑川 ゆき ] 製品画像:24位](https://thumbnail.image.rakuten.co.jp/@0_mall/book/cabinet/2033/9784592222033.gif?_ex=128x128)
![イラストでわかる建築設備 [ 山田信亮 ] 製品画像:23位](https://thumbnail.image.rakuten.co.jp/@0_mall/book/cabinet/3482/9784816353482.jpg?_ex=128x128)
![医者が考案した たんぱく質をたっぷりとる長生きスープ [ 土田隆 ] 製品画像:22位](https://thumbnail.image.rakuten.co.jp/@0_mall/book/cabinet/4168/9784776214168_1_3.jpg?_ex=128x128)
